OVERVIEW
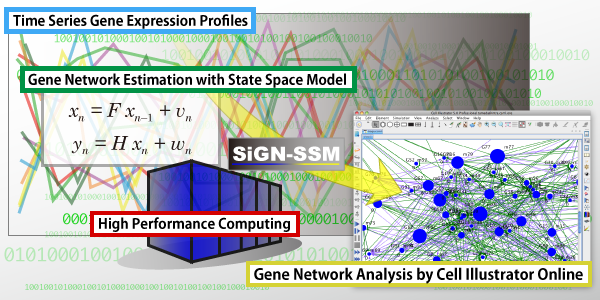
SiGN-SSM(サイン-SSM) は時系列遺伝子発現データから遺伝子ネットワークを推定するためのオープンソースソフトウェアです. SiGN-SSMは状態空間モデル(SSM: State Space Model) と呼ばれる動的な統計モデルを時系列の多変量観測データから推定します. 状態空間モデルは,核となる動的システムを記述する「モジュール」と, モジュールから各遺伝子への対応付けにより, 細胞内遺伝子発現の動的な変化および遺伝子間の依存関係をモデル化します. SiGN-SSM は様々な並列実行環境を用いて並列動作することが可能で, 通常の PC に搭載されたマルチコア CPU による並列実行から PC クラスタや 超並列スーパーコンピュータを用いた数百〜数千並列の動作に対応しています. SiGN-SSM は遺伝子間の動的な依存関係を解析するだけでなく, 時系列データを用いた有意に発現差のある遺伝子抽出も可能です.
SiGN-SSM は GNU AFFERO GENERAL PUBLIC LICENCE (GNU AGPL) version 3 のもとで配布されているオープンソースソフトウェアです. いくつかの OS/CPU 向けには,あらかじめコンパイルされたバイナリも用意されている他, ヒトゲノム解析センターのスーパーコンピュータ, および次世代スーパーコンピュータ「京」(2012年完成予定) にはあらかじめバイナリがインストールされており,ユーザが自由に使用することが可能です. SiGN-SSM のいくつかの機能はこれらのスーパーコンピュータだけの限定機能になっています.
このソフトウェアは文部科学省新学術領域研究「システムがん」でも利用されています.
FEATURES
CONTENTS
ACKNOWLEDGEMENTS
SiGN-SSM は,理化学研究所 次世代計算科学研究開発プログラムで研究・開発している 「次世代生命体統合シミュレーションソフトウェアの研究開発プロジェクト (ISLiM)」 の元で開発されたものです. このソフトウェアは TRANS-MNET として公開されているソフトウェアを基に改良したものです. SiGN-SSM の開発に必要な計算機資源は,ヒトゲノム解析センターのスーパーコンピュータ, 及び理化学研究所のスーパーコンピュータ RICC から提供されました.